L’évolution aviaire par la génomique
Fruit de quatre années de travail de près de 80 laboratoires à travers le monde, l’arbre phylogénétique aviaire, construit à partir du séquençage complet du génome de 48 espèces, renseigne sur les relations de parenté qui relient les oiseaux que nous connaissons aujourd’hui.
Roxane Tchernia - Publié le

Une trentaine de publications, dont huit dans l’édition du 12 décembre 2014 de la revue Science, près de 200 chercheurs, 20 nationalités, 80 laboratoires, des super-ordinateurs tournant à plein régime pendant plusieurs mois pour séquencer puis comparer les génomes de 48 espèces… C’est ce qu’il aura fallu pour enfin réactualiser et préciser l’arbre de parenté des oiseaux qui fournit un éclairage sur les relations ancestrales entre les différentes espèces que l’on connaît aujourd’hui.
C’est en 2010 que les choses ont véritablement commencé, quand Erich D. Jarvis (neurobiologiste à l’Institut médical Howard Hughes et à l’université Duke, aux États-Unis) et Thomas Gilbert (professeur au Centre de géogénétique du Muséum d’histoire naturelle au Danemark) ont réussi à convaincre l’Institut de génomique de Pékin (BGI pour Beijing Genomics Institute) de réaliser le séquençage du génome de 43 espèces d’oiseaux. Jusqu’à présent, les arbres n’étaient établis qu’à partir de quelques gènes. « Nous croyions qu’un important jeu de données nous donnerait la réponse », raconte Bastien Boussau, chercheur CNRS au Laboratoire de biométrie et biologie évolutive (Université Lyon-1), qui a participé au projet.
Fin 2011, les génomes de 48 espèces sont séquencés. Il s’agit alors de construire un arbre phylogénétique basé sur la comparaison de l’ensemble des gènes identifiés chez les différentes espèces. Pourtant, ce premier arbre n’est guère convaincant : il n’est pas en cohérence avec l’histoire évolutive connue. « En fait, il fallait adapter les modèles statistiques utilisés et les modifier en même temps que nous changions d’échelle », explique Bastien Boussau. En plus d’ajuster leurs outils d’analyse bio-informatique, les chercheurs se sont donc appuyés sur des principes de génétique des populations pour reconstruire un arbre à partir des données génomiques.
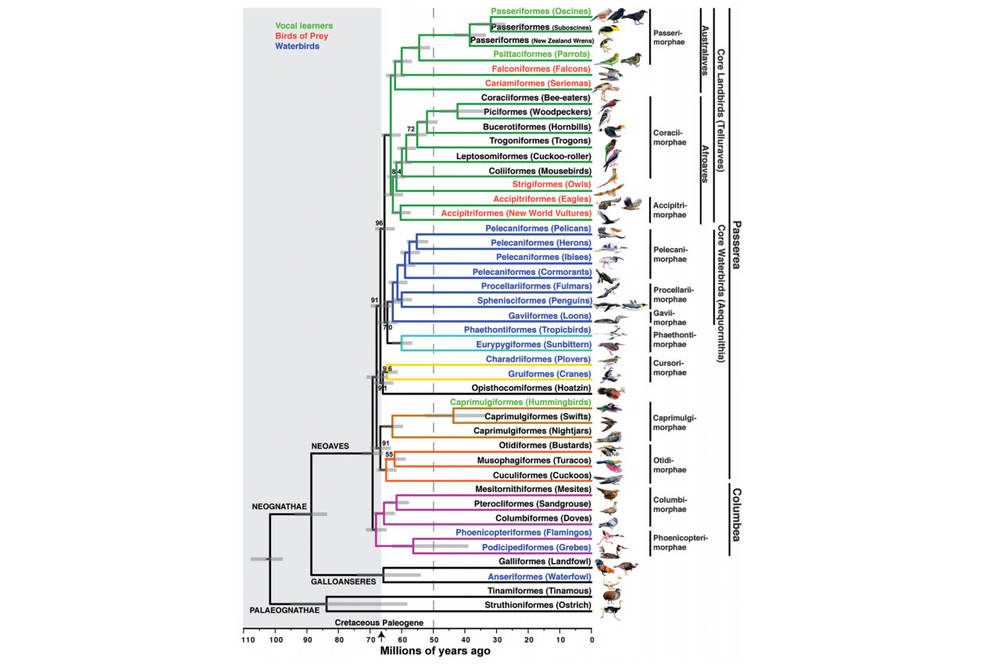
Le « Big Bang » de l’évolution aviaire
La diversification rapide des oiseaux à partir de la limite entre le Crétacé et le Tertiaire – marquée par l’extinction des grands dinosaures il y a 66 millions d’années – complique l’exercice classique de classification. En effet, la séparation des diverses branches du groupe aviaire s’étant produite en peu de temps – à l’échelle de l’évolution –, il y a peu de critères de spécification sur lesquels s’appuyer. L’approche génomique complète permet de dépasser cette barrière.
Les résultats publiés dans Science indiquent, par exemple, qu’un mécanisme génétique similaire est impliqué dans l’apprentissage vocal du chant chez les oiseaux et du discours chez l’Homme. Ils soulignent aussi que les mutations responsables de la perte de l’émail et de la dentine (supposée à l‘origine de la disparition des dents au profit du bec chez les oiseaux) seraient apparues il y a 116 millions d’années.
L’analyse des données aide également à mieux comprendre l’évolution des chromosomes sexuels (Z, W) du groupe aviaire. Une équipe de l’université de Californie, à Berkeley, a pu observer que contrairement au chromosome humain Y, le chromosome W, qui permet la différenciation sexuelle chez les oiseaux, posséderait encore beaucoup de gènes actifs.
Parmi ces résultats, certains permettent en fait de confirmer des hypothèses précédemment proposées, comme celles avancées par Shannon J. Hackett, conservatrice associée au Muséum d’histoire naturelle de Chicago, dans l’étude qu’elle a dirigée et publiée en 2008. Si son analyse s’appuyait sur moins de données génétiques que celle dont il est ici question – seulement 19 segments d’ADN au lieu du génome complet –, elle comparait, en revanche, beaucoup plus d’espèces – 169 contre 48.
Biologie et « big data »
Le travail qui a conduit à ce nouvel arbre de parenté des oiseaux illustre également l’arrivée du « big data » dans la recherche en biologie et la puissance potentielle de cet outil. Bastien Boussau, qui a participé à ce projet, craint même qu’il ne marque le début d’une « course à l’armement », dans laquelle des équipes de scientifiques, comme dans une sorte de compétition, proposeront successivement de nouveaux arbres de parenté des êtres vivants basés sur un nombre de plus en plus important de génomes séquencés. Il met aussi en garde contre l’un des problèmes posés par la manipulation d’un grand jeu de données : « La moindre erreur d’analyse va se répercuter avec beaucoup plus d’ampleur. C’est pourquoi il faut améliorer les modèles statistiques, et combiner ces méthodes avec d’autres, plus classiques ».

