La métagénomique à l’assaut de mondes inexplorés
Plancton océanique d’une incroyable diversité, flore intestinale aux vertus insoupçonnées, bactéries du sol productrices d’antibiotiques... autant de découvertes permises par une discipline en plein essor : la métagénomique.
Jean-Philippe Braly - Publié le
« L’ancêtre de nos cellules à noyau démasqué à 2 300 mètres de fond ! », « Sibérie : les sols gelés cachent un virus géant inconnu », « La flore intestinale impliquée dans une kyrielle de maladies », « L’expédition océanique Tara révèle une incroyable diversité planctonique »… Quel point commun relie ces récentes découvertes ayant fait les gros titres de la presse ? Réponse : aucune n’aurait été possible sans une discipline en plein boom dans les laboratoires du monde entier, la « métagénomique ».
Également nommée « génétique environnementale », cette discipline high-tech permet d’étudier la biodiversité de milieux ultra-complexes dont le moindre petit échantillon peut contenir des millions... voire des milliards de microorganismes. C’est par exemple le cas des océans, des sols... ou bien encore de notre tube digestif ! Dans ces conditions, impossible d’aller isoler chaque bactérie pour l’étudier au microscope... d’autant que plus de 99 % sont réfractaires à toute mise en culture au laboratoire ! Voilà pourquoi, les microbiologistes parlent de « matière noire microbienne ». À la croisée des chemins entre génétique, informatique et écologie, la métagénomique met en œuvre une tout autre stratégie : faire directement parler l’ADN de ces myriades de microorganismes.

Code barre génétique
Mais comment diable s’y prennent les chercheurs pour faire parler ces masses d’ADN ? On s’en doute, il faut d’abord réussir à extirper l’ADN des microorganismes contenu dans les échantillons prélevés dans le milieu complexe à étudier. Pour cela, plusieurs armes sont déployées : techniques chimiques ou enzymatiques pour faire éclater les bactéries, traitements physiques de broyage pour les sols, de filtration pour récupérer l’ADN de virus dans les eaux, etc. « Cette première étape doit encore être améliorée, concède toutefois Pascal Simonet, responsable de l’équipe “Génomique microbienne environnementale” au Laboratoire Ampère de l’École Centrale de Lyon. Par exemple, aujourd’hui, on ne parvient à récupérer que 20 % maximum de l’ADN d’un sol. »

À partir de cette « soupe d’ADN », la métagénomique offre plusieurs options. Nommée « metabarcoding », une nouvelle méthode en plein essor permet par exemple de dresser l’inventaire des espèces présentes, juste en amplifiant puis en séquençant quelques gènes bien particuliers dits « ribosomiques ». Les généticiens ont en effet découvert que la composition de ces gènes – leur « séquence » – est propre à chaque espèce... une sorte de code-barres génétique ! « Après la découverte d’une incroyable diversité du plancton océanique grâce à cette technique par l’expédition Tara, nous lançons PlanktonPlanet, un projet de metabarcoding citoyen des océans impliquant tous les navigateurs qui le souhaitent ! », s’enthousiasme Colomban de Vargas, chercheur CNRS à la Station Biologique de Roscoff.
Analyse massive
Autre stratégie aujourd’hui permise par la métagénomique : séquencer la totalité de l’ADN tiré des échantillons, les experts parlent de « séquençage massif ». Une prouesse désormais possible grâce à l’avènement de séquenceurs haut débit à la puissance incomparable. Rien qu’un chiffre pour s’en convaincre : certains peuvent séquencer plusieurs dizaines de milliards de « nucléotides » (les briques élémentaires de l’ADN)... par jour ! Pour se faire une idée de ce que cela représente, rappelons que la totalité du génome humain n’en contient « que » trois milliards... Les myriades de séquences génétiques ainsi obtenues vont ensuite ainsi être traitées par des logiciels informatiques dits de « Big Data ». Cette avalanche de données nécessite en effet des outils informatiques de plus en plus puissants en termes de capacité de stockage, et de puissance de calcul pour les interpréter. « Pour que la métagénomique puisse faire toute la lumière sur cette matière noire microbienne, nous avons désormais besoin d’une véritable cyber-infrastructure dotée de supercalculateurs aussi puissants que ceux utilisés par les astrophysiciens ou les météorologistes, et d’une véritable coopération internationale », lance Pascal Simonet. Pour l’heure les outils informatiques utilisés permettent déjà de comparer les séquences obtenues par les séquenceurs haut débit, avec celles stockées dans ces immenses bases de données génétiques dont on connaît déjà les fonctions.

Et si les séquences s’avèrent inconnues ? Qu’à cela ne tienne ! En effet, ces logiciels sont aussi capables d’émettre des hypothèses sur leurs fonctions et leurs porteurs, sur la base de leurs similarités avec des séquences connues stockées dans ces bases de données. Pour confirmation, les chercheurs peuvent ensuite créer des « clones », autrement dit des bactéries de laboratoire dans lesquelles on insère la séquence génétique choisie pour qu’elles l’expriment. Une étape nommée « criblage métagénomique ». C’est ainsi qu’on découvre qu’une séquence jusqu’ici inconnue code par exemple une molécule antibiotique, une enzyme capable de dégrader tel ou tel composé, etc. Au Laboratoire Ampère de l’École Centrale de Lyon, l’équipe de Pascal Simonet s’est ainsi constitué une collection de plus de deux millions de clones bactériens ! « Chacun d’entre eux contient un fragment d’ADN d’une parmi ces milliards de bactéries inconnues qui compose le microbiote du sol et que permet donc d’étudier et même d’exploiter la métagénomique », indique le chercheur.
Des antibiotiques sous nos pieds !
Appliquées aux sols – dont un seul gramme peut contenir jusqu’à un milliard de microorganismes – ces recherches métagénomiques font déjà des merveilles. Elles ont notamment permis de découvrir des bactéries productrices de nouveaux antibiotiques. Enfoui juste là sous nos pieds, ce gisement d’antibiotiques naturels est inespéré face au problème de santé publique majeur posé par la résistance croissante aux antibiotiques aujourd’hui disponibles. « Une récente étude sur un site de production d’antibiotiques en Inde a révélé la présence de bactéries résistantes à 35 antibiotiques différents ! », confirme Pascal Simonet.
Dans la même veine, une récente étude métagénomique sur 467 stations de métro new-yorkaises a révélé que 48 % des séquences génétiques prélevées ne correspondaient à aucun microorganisme connu... et que 28 % des quelque 1 700 groupes bactériens isolés résistaient à au moins un antibiotique courant ! Depuis, un projet nommé MetaSUB ambitionne d’étudier tout le matériau génétique qu’on pourra trouver dans les transports publics de 16 grandes villes dans le monde. Les résultats pourraient améliorer le suivi des épidémies urbaines, de l’impact de pollutions ou d’événements climatiques... ou bien encore la lutte contre le bioterrorisme.
Des sols gelés de Sibérie... aux grands fonds marins norvégiens
Fin 2015, dans un sol gelé en permanence (permafrost) au nord-est de la Sibérie, la métagénomique a carrément permis de dénicher un virus géant dont la forme, le métabolisme et le mode de réplication ne ressemblent à rien de connu ! De quoi inquiéter les scientifiques qui craignent la résurgence de virus pathogènes dans cette région du monde dont l’accessibilité et l’exploitation industrielle sont facilitées par le changement climatique... En termes de milieux difficiles d’accès, la métagénomique s’attaque aussi aux grands fonds marins. C’est ainsi qu’en mai 2015, des chercheurs norvégiens et suédois ont publié une découverte étonnante dans la célèbre revue Nature.
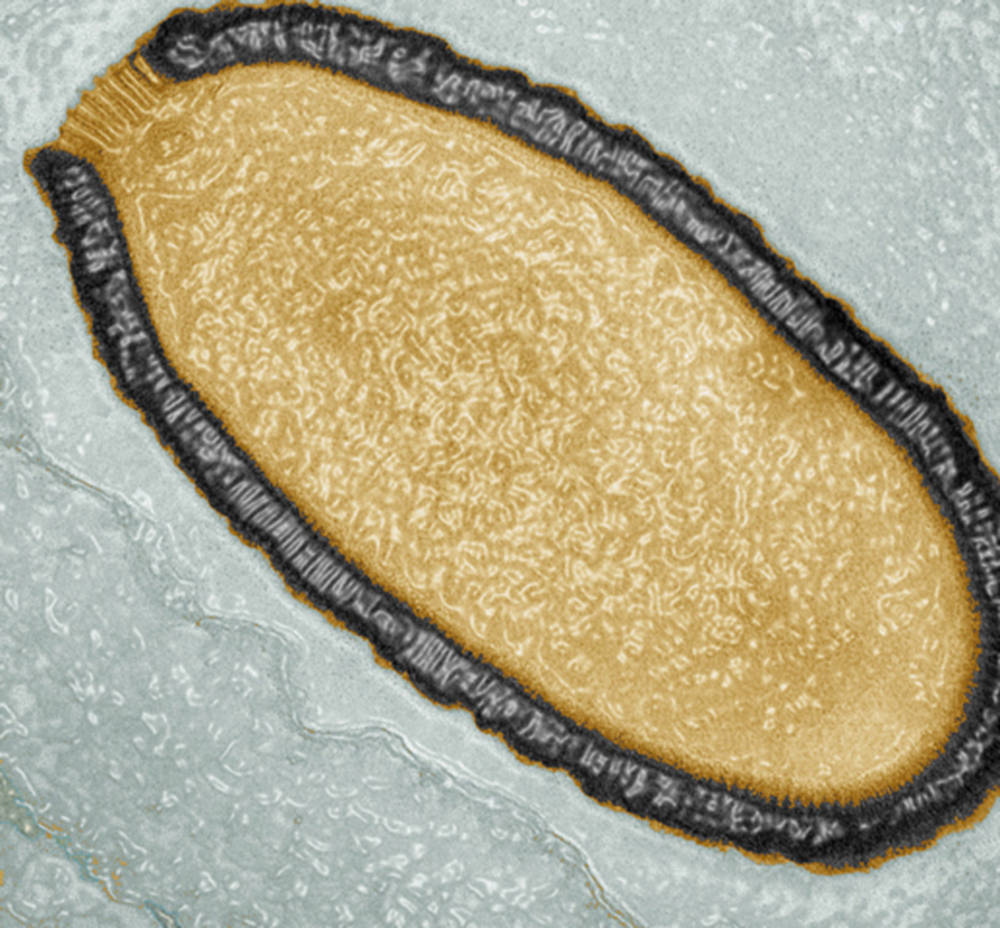
En analysant par métagénomique les sédiments d’une source hydrothermale située à plus de 2 300 m de fond entre le Groenland et la Norvège nommée « château de Loki » – cette équipe a mis la main sur un tout nouveau groupe d’archées, ces micro-organismes primitifs unicellulaires dépourvus de noyau. Baptisé Lokiarchaeota, ce groupe possédait de nombreux gènes caractéristiques des cellules à noyau (eucaryotes). De quoi accréditer la thèse selon laquelle les archées constitueraient le chaînon qui manquait entre organismes procaryotes et organismes eucaryotes apparus il y a plusieurs milliards d’années... et dont nous sommes les fiers descendants ! Mais pas besoin d’aller aussi loin qu’en Sibérie ou aussi profond que le château de Loki pour découvrir une biodiversité insoupçonnée. C’est aussi le cas dans les lacs où la métagénomique déniche de nombreux virus jusqu’ici inconnus dotés de gènes jamais observés auparavant. « La métagénomique a déjà fait des découvertes passionnantes sur le monde viral : recombinaisons entre virus à ADN et à ARN, virus porteurs d’ADN bactérien, gènes viraux jusqu’ici considérés propres aux bactéries pour oxyder le soufre, etc. », confirme François Enault, bio-informaticien spécialiste des virus aquatiques à l’université de Clermont-Ferrand.
Une terra incognita... dans notre ventre !
L’autre grande terra incognita à haute biodiversité désormais explorée par la métagénomique se cache... dans notre ventre ! Et oui, selon une nouvelle estimation, notre flore intestinale est riche de 30 000 milliards de bactéries... soit 1,3 fois plus que les propres cellules de notre corps.
Au fil des nombreuses études déjà menées sur des personnes saines ou souffrant de diverses pathologies, les chercheurs sont allés de surprises en surprises. Ils ont ainsi découvert que ces milliards de bactéries intestinales ne sont pas impliquées que dans la digestion... mais aussi dans le fonctionnement du métabolisme, du système immunitaire, ou bien encore du système nerveux ! Voilà pourquoi on suspecte aujourd’hui l’implication de déséquilibres de la flore intestinale dans toute une série de maladies : obésité, diabète, allergies, pathologies cardiovasculaires et inflammatoires... De quoi inciter le développement de stratégies capables de rétablir une flore saine : probiotiques, régimes spécifiques, médicaments... Plus étonnant, certains médecins envisagent très sérieusement de transplanter des matières fécales dotées d’une flore saine aux sujets malades ! Déjà expérimentée aux Pays-Bas sur 16 patients atteints d’une grave infection intestinale, cette technique de « greffe fécale » a guéri 13 d’entre eux... Toutefois, pour éviter tout problème éthique à la possible transmission de virus pathogènes, l’avenir de cette technique devrait plutôt passer par l’auto-transplantation (flore saine conservée puis transplantée à son propriétaire devenu malade).
Toujours plus loin
Mais à l’avenir, les découvertes pourraient être encore plus impressionnantes. En effet, les scientifiques espèrent bientôt pouvoir reconstituer la composition du génome de chaque bactérie initialement présente dans l’échantillon étudié par métagénomique ! Une prouesse informatique réservée, pour l’instant, à des environnements à la biodiversité restreinte : une équipe de Strasbourg y est par exemple parvenue sur des sédiments sortant de bassins de mines très acides… On l’aura compris, la métagénomique n’a pas fini de faire les gros titres !
En savoir plus
« La Métagénomique – Développements et futures applications », de Marie Christine Champomier-Vergès et Monique Zagorec aux Éditions Quae, 2 015.

